Kapitel 2 Introduktion til R
R er et program og programmeringssprog designet til statistiske udregninger og grafik.1 Vi bruger R, fordi det er de facto standarden indenfor statistisk software, fordi det er et af de stærkeste statistiske analyseværktøjer på markedet, fordi det gør det let at reproducere sine resultater, og fordi R nyder godt af et stort og dynamisk brugermiljø. Det betyder, at mange nyudviklede visualiserings- og analysemetoder publiceres med kode til R, så alle brugere kan anvende disse nye metoder uden at skulle programmere dem selv.

2.1 RStudio
R fungerer ved at skrive kommandoer, som efterfølgende udføres. Det anbefales at bruge en editor som RStudio til at skrive og holde styr på programmerne, lave reproducerbar forskning, håndtere grafik og vise Rs hjælpesider. Når både R og RStudio er installeret, er det kun nødvendigt at starte RStudio.2
RStudio er opdelt i fire mindre vinduer (figur 2.1). Øverste venstre vindue er en teksteditor, hvor R programmerne skrives.3 Kommandoerne i editoren udføres først, når man trykker “Run” på menubjælken (så bliver alle kommandoerne i editoren udført i rækkefølge), eller alternativt vil ( på Mac) udføre kodelinjen, som cursoren står på i editoren. Markeres flere linjer sendes de alle til R og eksekveres i rækkefølge.
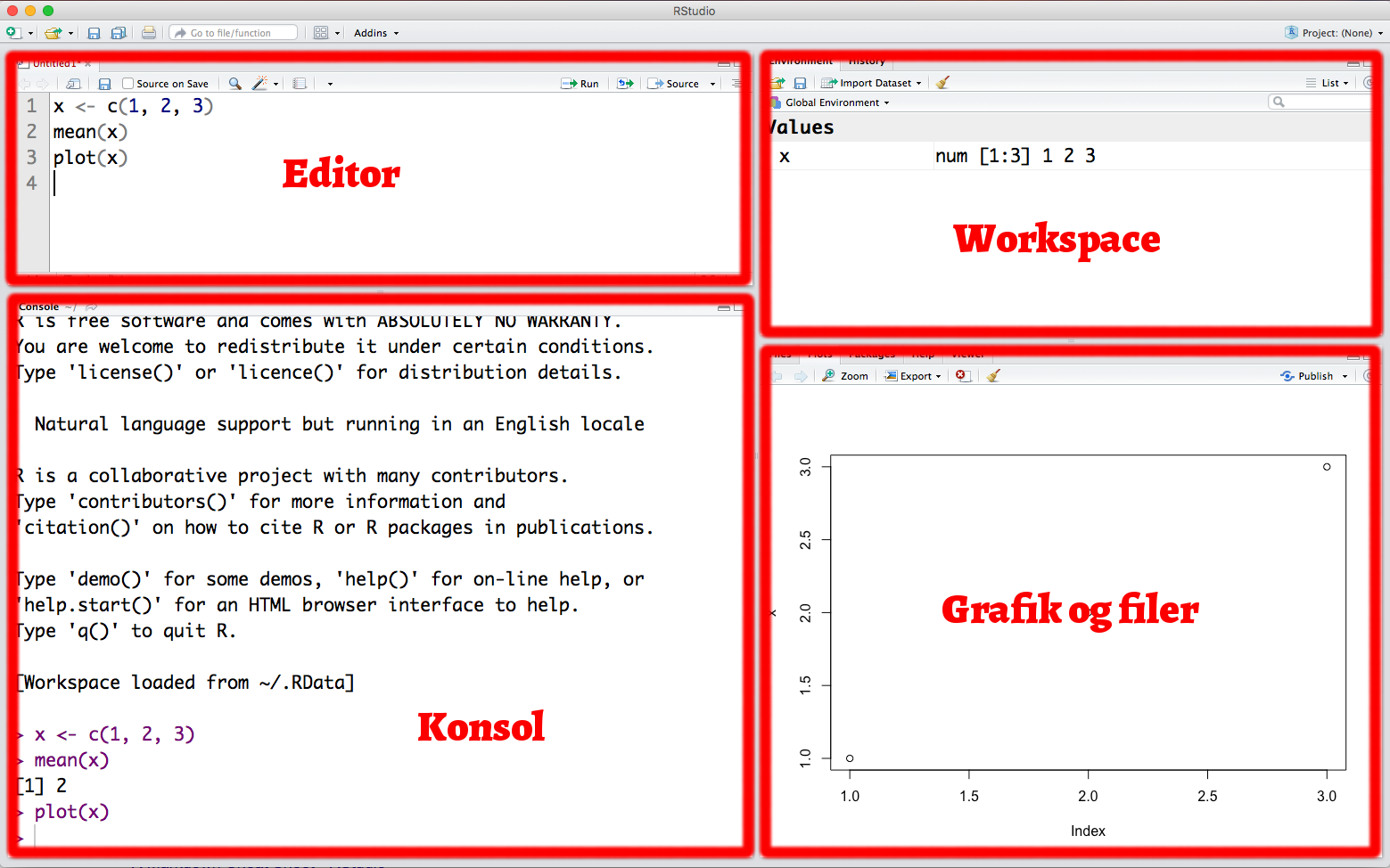
Figure 2.1: RStudios brugergrænseflade er inddelt i 4 mindre vinduer: Øverst til venstre findes editoren, hvor man kan skrive, gemme og hente sit R program. Nederst til venstre er konsollen, hvor man har direte adgang til R, og hvor resultatet af udregningerne vises. Øverst til højre vises det nuværende workspace og historikken, og nederst til højre findes en filoversigt, og det er også her grafik, figurer og hjælpesider bliver vist.
I nederste venstre vindue af RStudio findes konsollen, hvor selve R kører. Her vises resultatet af udregningerne. Når kommandoer overføres fra editorvinduet som beskrevet ovenfor, så bliver de i praksis kopieret ned i konsollen og kørt linje for linje.
Øverste højre vindue viser Rs workspace — en oversigt over objekterne i Rs hukommelse. Fanebladet “History” viser historikken over alle de kommandoer, der hidtil har været kørt. Markeres en eller flere af linjerne i historikken så sendes de til konsollen og bliver kørt igen, hvis der dobbeltklikkes på dem. Det er muligt at gemme hele historikken i en fil, så man den vej igennem kan dokumentere og genskabe de kommandoer, der har været kørt.
Grafik bliver vist nederst til højre. Herfra er det muligt at eksportere figurer, så de kan anvendes i andre programmer. Det er også her, at Rs hjælpesider vises, og hvor en filoversigt over Rs arbejdsbibliotek kan ses.4
2.1.1 Tricks når man arbejder med R
Der er en række tricks, der er værd at kende til i arbejdet med R.
I konsollen viser R en prompt,
>, når programmet er klar til at modtage kommandoer. Får man ikke færdiggjort en selvstændig kommando skifter prompten til+, og det er muligt at færdiggøre ordren. afbryder indtastningen og man kommer tilbage til>.I konsollen bladrer og op og ned i gamle kommandoer. Dermed kan det undgås at skulle skrive gamle kommandoer helt forfra.
Funktioner i R angives altid med parenteser og har formen
funktionsnavn(argument1, argument2, ...), hvor det er muligt at give nul eller flere argumenter afhængigt af den konkrete funktion. På den måde kan man altid skelne mellem almindelige objekter (uden parentes) og funktioner.5RStudio har “auto-completion”: når man starter med at taste i editoren eller konsollen foreslår RStudio, hvad der kunne stå. Trykkes færdiggør RStudio resten af navnet på objektet/funktionen.
2.2 Installation og brug af pakker
Der findes mere end 15000 pakker, som udvider R med nye funktioner og
datasæt. I RStudio installeres nye pakker via menupunktet Tools > Install Packages (se figur 2.2, hvor
pakken isdals installeres). Alternativt kan en pakke installeres
direkte med funktionen install.packages().
install.packages("isdals") # Installér pakken isdals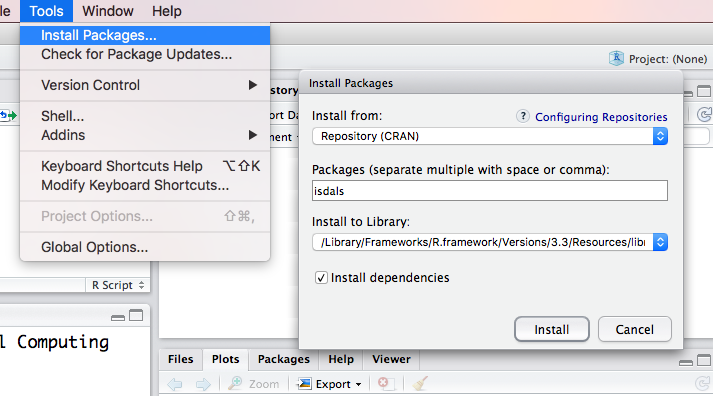
Figure 2.2: Installation af nye pakker gennem RStudios brugergrænseflade foregår via menupunktet Tools > Install Packages. Her installeres pakken isdals fra CRAN, der er Rs officielle netværk af tilgængelige pakker.
Det er kun nødvendigt at installere en pakke én gang. For at bruge funktionerne eller datasættene fra en pakke skal den indlæses, hvilket gøres med
For at spare på computerens hukommelse indlæses installerede pakker
kun, når brugeren ønsker det. Indlæsningen foregår med kommandoen
library(), hvilket er nødvendigt hver gang R startes op, hvis den
pågældende pakke skal bruges.6
# Få adgang til data og funktioner fra pakken isdals
library("isdals")Datasæt gøres tilgængelige med kommandoen data(). Hvis data ligger
i en pakke skal pakken først indlæses ved hjælp af library().
library(isdals) # Hent pakken isdals
data(aids) # Indlæs datasættet aids
head(aids) # Vis de første 6 linjer## year cases deaths
## 1 1981 339 130
## 2 1982 1201 466
## 3 1983 3153 1511
## 4 1984 6368 3526
## 5 1985 12044 6996
## 6 1986 19404 121832.3 R som lommeregner
R fungerer som en helt almindelig lommeregner. Tastes et regneudtryk ind i editoren (øverste venstre vindue), hvorefter det markeres så vil ( på Mac) automatisk kopiere koden ned i konsollen og køre den.
1 + 5 # Alt efter # er en kommentar: udregn 1 + 5## [1] 6Svaret på 5+1 aflæses til
6.7 De fire almindelige regnearter: +, -, * og / fungerer som
normalt ligesom ^ (opløftning i potens) samt naturligvis
parenteser.
3 * (1 + 5) / (4 + 2^2) # Langt regneudtryk## [1] 2.25Udregningen af \(\frac{3\cdot(1+5)}{4 + 2^2}\) er 2.25.
Resultatet kan gemmes i et objekt, så det kan
genbruges. R bruger “pilen” <- til at gemme et objekt.8 Navnet på det gemte objekt kan vælges næsten frit:
det skal dog starte med et bogstav og må ikke indeholde +, -, *,
/, ^, ;, (, ) og komma.
y <- 5 # Gem værdien 5 i variablen y
y # Udskriv værdien af y## [1] 5y * 2 # Udregn 2 * værdien af indholdet af y## [1] 10Mere komplicerede matematiske operationer kan udregnes ved hjælp af funktionerne vist i tabel 2.1.
| Funktion | Forklaring | Eksempel |
|---|---|---|
abs(x) |
Numerisk (absolut) værdi | abs(-3) = 3 |
sqrt(x) |
Kvadratrod | sqrt(9) = 3 |
a^x |
\(a\) i \(x\)te potens | 4^2 = 16 |
log(x) |
Naturlig logaritme | log(10) = 2.303 |
log10(x) |
Titalslogaritme | log10(10) = 1 |
log(x, base=2) |
Totalslogaritme | log(10, base=2) = 3.322 |
exp(x) |
Eksponentialfunktionen | exp(1) = 2.718 |
R skelner mellem store og små bogstaver, så Y og y er to
forskellige objekter, hvilket kan give anledning til overraskelser.9
Y # Et ukendt objekt## Error in eval(expr, envir, enclos): object 'Y' not foundR indeholder en ret veludbygget dokumentation, og hjælpesiden til
en funktion eller et datasæt kan ses med
funktionen help().
help(sqrt) # Hent hjælpesiden for funktionen sqrtOvennævnte viser hjælpesiden for sqrt() og giver en grundig
forklaring af funktionen og dens tilhørende argumenter.
Kender man ikke navnet på den relevante R funktion, kan help.search() søge hjælpesiderne igennem.10
help.search("box plot") # Find funktioner om "box plot"Dette viser en liste over de funktioner, der omtaler “box plot”. Forhåbentlig giver listen en ide om, hvilken R funktion, der måtte være relevant.
2.4 Vektorer
Vektorer er en af Rs helt store styrker. En vektor er en samling af elementer af samme type — eksempelvis tal eller tekst.
En vektor kan laves med funktionen c() (for
combine eller concatenate), der “klistrer” elementer sammen.
hojde <- c(1.65, 1.79, 1.62, 1.87) # Højde for 4 patienter
hojde # Udskriv højderne## [1] 1.65 1.79 1.62 1.87vaegt <- c(55.2, 89.7, 49.8, 92.1) # Vægt for samme 4 personer
vaegt # Udskriv vægtene## [1] 55.2 89.7 49.8 92.1Fordelen ved vektorer er, at R kan lave samme udregning for hvert enkelt element i vektoren. Body-mass index (BMI)11 for alle fire personer udregnes ved
bmi <- vaegt / (hojde^2) # Udregn BMI og gem som bmi
bmi # Udskriv bmi## [1] 20.27548 27.99538 18.97577 26.33761hvilket direkte giver resultaterne for alle fire patienter. Bemærk, at
R regner elementvis, så det første element i hojde bruges sammen med
det første element i vaegt og så videre. Rækkefølgen på målingerne
er altså vigtig og skal passe sammen.
Hvis vektorerne i en udregning ikke har samme længde, så undersøger R først, om længden af den korte vektor går op i længden af den lange vektor. Hvis det er tilfældet bliver den korte vektor gentaget indtil den har samme længde som den lange vektor.12 Hvis vi eksempelvis hellere ville have målt højderne i cm i stedet for i meter så skal vi gange alle værdierne med 100.
hojde * 100 # Gang hver enkelt højdeværdi med 100## [1] 165 179 162 187Denne udregning går godt, fordi hojde er en vektor af længde 4, og tallet
100 opfattes om en vektor af længde 1. Da 1 går op i 4 gentages
værdien 100 4 gange indtil de to vektorer er lige lange, og så arbejder R
elementvis igen.
Vektorer er især nyttige i kombination med de indbyggede matematiske
funktioner, da disse accepterer vektorer som input. For eksempel tager
log() den naturlige logaritme af hvert element i en vektor.
log(hojde) # Tag logaritmen til hvert element i hojde## [1] 0.5007753 0.5822156 0.4824261 0.6259384Nogle funktioner bruger hele vektoren til at udregne nye
resultater. Eksempelvis udregnes gennemsnittet af de fire personers
BMI ved at give vektoren til funktionen mean().
mean(bmi) # Udregn gennemsnittet af alle værdierne i bmi## [1] 23.39606R deler outputtet op i flere linjer for lange lange vektorer, der ikke
kan udskrives på en enkelt linje. Input kan også splittes over flere
linjer. Så skifter Rs inputprompt midlertidigt fra > til + så det
er tydeligt, at man skriver videre på linjen ovenfor.
langvektor <- c(1, 3, 5, 7, 9, 2, 4, 6, 8, 10, 20, 21, 22,
23, 24, 25, 26, 27, 28, 29, 30, 100, 101,
102, 103, 104, 105, 106, 107, 108, 109)
langvektor## [1] 1 3 5 7 9 2 4 6 8 10 20 21 22 23 24 25 26 27 28
## [20] 29 30 100 101 102 103 104 105 106 107 108 109Vektorer behøver ikke indeholde tal, men kan for eksempel også bestå af
tekst eller logiske værdier (sandt eller falsk — benævnt TRUE eller
FALSE i R).
v2 <- c("Bamse", "Kylling", "ælling") # En vektor af tekst
v3 <- c(TRUE, FALSE, FALSE) # En logisk vektor
v4 <- c(1, 2, 5) # En vektor af tal
v2 # Udskriv v2## [1] "Bamse" "Kylling" "ælling"v3 # Udskriv v3## [1] TRUE FALSE FALSEv4 # Udskriv v4## [1] 1 2 52.4.1 Indicering i vektorer
Der kan henvise til specifikke elementer i én vektor med en anden vektor med de relevante indices i skarpe parenteser.
v2[c(2, 3)] # Udtræk element nummer 2 og 3 fra v2## [1] "Kylling" "ælling"Indicering kan også bruges til at ændre specifikke elementer.
v2[3] <- "Ælling" # Skift element 3 til at starte med stort
v2 # Udskriv den nye v2## [1] "Bamse" "Kylling" "Ælling"Ved indicering kommer elementerne ud i den rækkefølge de bliver refereret. Elementernes rækkefølge kan byttes rundt ved at ændre rækkefølgen på indices.
v2[c(2, 3, 1)] # Udskriv v2 i ny rækkefølge## [1] "Kylling" "Ælling" "Bamse"Bemærk at v2 ikke ændres med ovennævnte kommando, da kommandoen blot
udskriver den vektor, hvor der er byttet rundt på
rækkefølgen. Resultatet skal gemmes, hvis det skal bruges i anden
sammenhæng:
v5 <- v2[c(2, 3, 1)] # Lav v5 med ny rækkefølge af v2
v5## [1] "Kylling" "Ælling" "Bamse"En logisk vektor af samme længde som den oprindelige vektor kan også bruges som indeks. Så udvælges netop de elementer, hvor den logiske vektor er sand.
v2[v3] # Udskriv de elementer i v2, hvor v3 er sand## [1] "Bamse"Denne funktionalitet er ekstra nyttig, hvis man gerne vil udvælge elementer på baggrund af værdierne i en anden vektor.
v2[v4 < 5] # Vælg værdier af v2, hvor v4 er mindre end 5## [1] "Bamse" "Kylling"Element 1 og 2 er netop de tilfælde, hvor værdien af v4 er mindre
end 5.
I R bruges en lidt speciel notation med to lighedstegn, når det skal vurderes om to objekter er ens.
v2[v4==2] # Udvælg når v4 er 2. Bemærk to lighedstegn!## [1] "Kylling"2.5 Data frames
Data frames er Rs betegnelse for et datasæt, der indeholder en samling af
vektorer (variablene i datasættet), der alle har samme længde.
En data frame laves ved at sætte vektorer af samme længde sammen ved
hjælp af funktionen data.frame(), og det er muligt at give vektorerne
nye navne, når datasættet laves.

Figure 2.3: Man kan tænke på en data frame som et firkantet område af et regneark, hvor hver række svarer til en observation, en person eller en registrering, og hvor hver søjle svarer til en variabel.
DF <- data.frame(navn=v2, # Lav data frame
bjørn=v3, # Med tre variabel og
v4) # giv nye navne til to
DF # Udskriv data frame## navn bjørn v4
## 1 Bamse TRUE 1
## 2 Kylling FALSE 2
## 3 Ælling FALSE 5Variablene i en data frame eksisterer kun inde i selve data framen. Det er ret smart, for så kan samme variabelnavn benyttes i flere forskellige data frames uden at de overskriver hinanden.
DF2 <- data.frame(navn=c("Palle", "Polle", "Ruth"),
v4=c(1,2,3))
DF2 # Udskriv data framen. Bemærk variablene navn og v4## navn v4
## 1 Palle 1
## 2 Polle 2
## 3 Ruth 3DF # Den gamle data frame. Har også variablene navn og v4## navn bjørn v4
## 1 Bamse TRUE 1
## 2 Kylling FALSE 2
## 3 Ælling FALSE 5Da variable i en data frame kun kan ses inde i den data frame, hvor de
er defineret, er det nødvendigt også at angive navnet på den relevante
data frame, når variablen benyttes i andre sammenhænge. Eksempelvis ved
R ikke, om variablen v4 henviser til den pågældende variabel i DF,
eller v4 fra DF2. Referencer til variable inde i en data frame
opnås ved at bruge $ mellem navnet på data framen og
variablen.13
DF$v4 # Hent v4 fra DF## [1] 1 2 5DF2$v4 # Hent v4 fra DF2## [1] 1 2 3Nye variable kan tilføjes til en eksisterende data frame ved bare at definere nye variable. Eneste krav er, at nye variable skal have samme længde som de øvrige variable.14
DF$sko <- c("ja", "nej", "nej") # Lav variabel sko i DF
DF## navn bjørn v4 sko
## 1 Bamse TRUE 1 ja
## 2 Kylling FALSE 2 nej
## 3 Ælling FALSE 5 nej2.5.1 Indicering i data frames
Data frames kan opfattes som firkantede regneark (se figur 2.3), og specifikke elementer kan refereres ved at bruge to indices svarende til rækker og søjler.
# Hent anden observation fra tredie variabel
# svarende til række 2 søjle 3 i DF
DF[2, 3]## [1] 2Hele rækker og søjler udtrækkes ved at undlade at angive værdier for henholdsvis søjler og rækker.
DF[3,] # Hent hele række 3## navn bjørn v4 sko
## 3 Ælling FALSE 5 nejDF[,c(2, 4)] # Hent søjler 2 og 4## bjørn sko
## 1 TRUE ja
## 2 FALSE nej
## 3 FALSE nejLigesom for vektorer kan indicering bruges til at indsætte nye værdier for enkeltobservationer, rækker eller søjler.
DF$v4[1] <- 10 # Sæt første element af v4 i DF til 10
DF[,2] <- c(TRUE, TRUE, FALSE) # Erstat hele 2. søjle
DF # Udskriv data framen## navn bjørn v4 sko
## 1 Bamse TRUE 10 ja
## 2 Kylling TRUE 2 nej
## 3 Ælling FALSE 5 nej2.6 Tabeller og matricer
En matrix er en “firkantet samling” af tal, hvilket kan bruges til at
repræsentere antalstabeller.15 I R laves matricer ved hjælp af matrix(), og
som input bruges en vektor af de tal, der skal fyldes ind i matricen,
og det ønskede antal rækker eller søjler.
m <- matrix(c(1, 2, 3, 4, 5, 6), # Matrix med tallene 1 2 3 4 5 6
2) # Inddel i to rækker
m## [,1] [,2] [,3]
## [1,] 1 3 5
## [2,] 2 4 6Det sidste tal, 2, i kaldet til matrix() ovenfor bestemmer antallet
af rækker, som tallene skal fyldes ind i. Bemærk at matricen fyldes op
søjlevis.16 Hvis antallet af
rækker ikke går op i antallet af tal i vektoren, så genbruger R
vektoren indtil matricen er fyldt helt ud, og skriver en advarsel.
m <- matrix(c(1, 2, 3, 4, 5), 2) # Matrix med for få tal## Warning in matrix(c(1, 2, 3, 4, 5), 2): data length [5] is not a sub-multiple or
## multiple of the number of rows [2]m## [,1] [,2] [,3]
## [1,] 1 3 5
## [2,] 2 4 1De fem første elementer i matricen fyldes op med værdierne 1 til 5, hvorefter vektoren genbruges forfra. Sidste element er derfor 1.
Matricer indiceres på samme måde som data frames. Elementet i 1. række og 3. søjle er
m[1, 3] # Udskriv element fra 1. række, 3. søjle## [1] 5m[1, 3] <- 8 # Erstat element 1, 3
m # Udskriv m## [,1] [,2] [,3]
## [1,] 1 3 8
## [2,] 2 4 12.7 Import af data
R kan importere data direkte fra de fleste dataformater og -baser. De simpleste formater kan indlæses direkte i R, mens mere komplicerede dataformater kræver, at en relevant pakke er installeret (se tabel 2.2 for en oversigt).
Filnavnet angives som første argument, men indlæsningsfunktionerne i
tabel 2.2 har lidt forskellige argumenter alt
afhængigt af dataformatet. Eksempelvis vil følgende kode indlæse
første ark fra Excelfilen forsøgsdata.xlsx.17
library("readxl") # Indlæs pakken readxl
minedata <- read_excel("forsøgsdata.xlsx", sheet=1)| Filformat (typisk filnavn) | Funktion | Pakke |
|---|---|---|
Tekstfil (.txt) |
read.table() |
|
CSV (.csv) |
read.csv() eller read.csv2() |
|
Excel (.xlsx eller .xls) |
read_excel() |
readxl |
SPSS (.sav eller .por) |
read_sav() eller read_por() |
haven |
SAS (.sas7bdat) |
read_sas() |
haven |
Stata (.dta) |
read_stata() |
haven |
JSON (.json) |
fromJSON() |
jsonlite |
Hjælp til at importere en række almindelige tekstformater kan findes
direkte i RStudio via menupunktet File > Import Dataset. Nedenfor
er vist indholdet af CSV-filen
patienter.csv.18
Navn;Alder;Status;BMI;Kon
Jensen;61;1;31,4;M
Hansen;26;0;23;M
Petersen;77;0;28,3;K
Jørgensen;53;1;24,6;K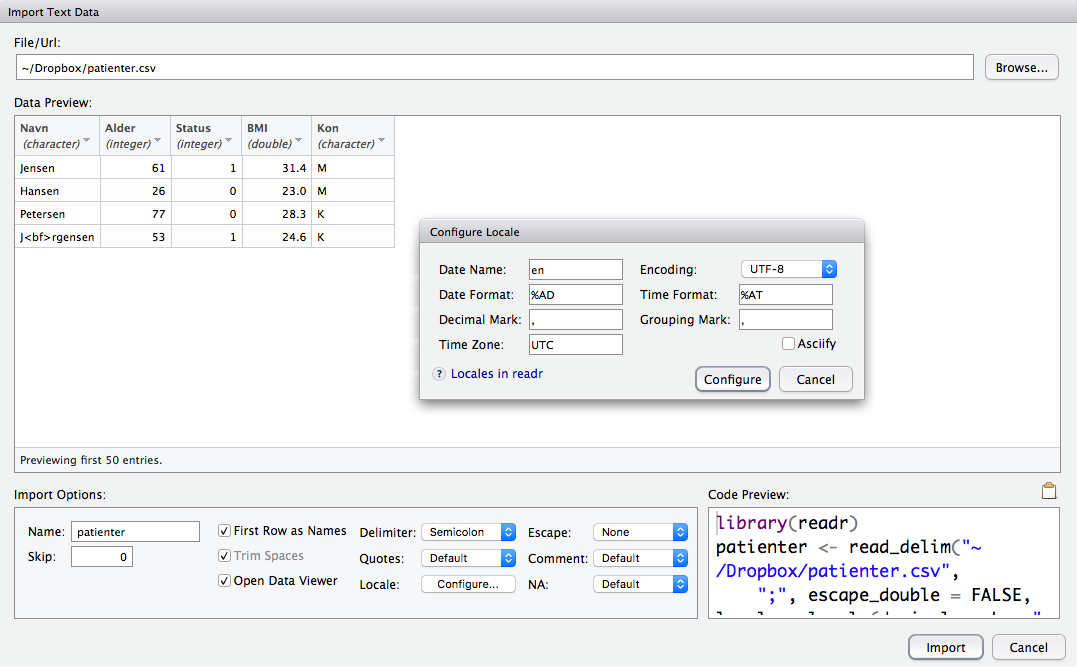
Figure 2.4: Import af data gennem RStudios File > Import Dataset > From CSV. Her importeres data fra tekstfilen patienter.csv, og nederst er markeret, at søjleadskiller er semikolon. Den lille dialogboks dukker op efter tryk på ‘Configure’, og her kan man sætte decimaladskiller. Vinduet ‘Data preview’ viser, hvordan R ser filen med de givne indstillinger, og at det ser fornuftigt ud. R finder de fire søjler med de korrekte formater.
Figur 2.4 viser dialogboksen, der
fremkommer, når man vælger File > Import Dataset > From CSV. Øverst
vælges den tekstfil, der skal indlæses, og nederst markeres, at
første linje i filen indeholder variabelnavnene (First Row as Names), og at variablene (søjlerne i filen) er adskilt med semikolon
(Delimiter).
Med knappen “Configure ..” fremkommer den lille dialogboks, og det angives, at decimaladskiller er komma (det bruges i
filen for variablen BMI) og ikke punktum, der er Rs almindelige
decimaladskiller.
Det store vindue “Data preview” viser, hvordan R ser den pågældende
fil med de givne indstillinger (figur 2.4)
Her indlæses filen som fire søjler svarende til de fire
variable i den oprindelige fil, og BMI er korrekt
indlæst som en double (en numerisk variabel) med de korrekte værdier.
Hvis indlæsningsindstillingerne er sat forkert bliver det klart i
dette vindue.
Nederst til højre fremgår de kommandoer, som R kører for at indlæse filen. Disse kommandoer bør kopieres over i editoren, så datafilen automatisk indlæses korrekt en anden gang.19
2.8 Databehandling med tidyverse
Data skal ofte tilpasses før de er klar til at blive
analyseret. Variable skal have det rigtige format, der skal laves nye
variabel, og datasættet skal måske omstruktureres. tidyverse er et
sæt af pakker til R, der gør databehandling meget lettere.
Funktionerne i tidyverse tager en data frame som første argument og
returnerer selv en data frame.20 Dermed kan funktionerne let
kombineres ved hjælp af operatoren |>, der kæder resultaterne fra
flere kommandoer sammen: resultatet af den første funktion bliver
brugt som input til den næste funktion.21 Funktionerne i tidyverse har desuden den
fordel, at det ikke er nødvendigt at referere til variable inde i en data
frame med $. Tabel 2.3 viser de mest
almindelige funktioner.

Figure 2.5: tidyverse introducerer funktionerne, pivot_longer() og pivot_wider(), der er nyttige at kende til, men som vi ikke vil komme nærmere ind på her. Funktionerne kan omstrukturere datasæt fra “wide format” til “long format” eller den anden vej. “Wide” datasæt optræder typisk, når der er gentagne målinger på samme forsøgsenhed. Et “long” datasæt indeholder den samme information, men de gentagne registreringer er spredt ud over flere rækker. Mange analysemetoder i R kræver data i “long” format.
| Funktion | Forklaring |
|---|---|
arrange() |
Sortér data |
fct_collapse() |
Slå kategorier af en faktor sammen |
filter() |
Vælg observationer (rækker) |
group_by() |
Split data op i undergrupper |
mutate() |
Ændre/lav nye variable |
pivot_longer() |
Kombiner søjler, så datasættet bliver “langt” |
pivot_wider() |
Tag to søjler (navne og værdier) og gør data “bredt” |
pull() |
Udtræk en variabel som en vektor |
select() |
Vælg søjler (variable) |
summarise() |
Opsummere data ud fra grupper |
ungroup() |
Fjern undergrupper |
Som et eksempel på databehandling med tidyverse bruges et datasæt om
behandling af diastolisk blodtryk hos 10 forsøgsdeltagere. De er alle
målt to gange: ved forsøgsstart og efter 4 uger (variablene start og 4 uger). Desuden er der en
række yderligere registreringer på forsøgsdeltagerne, hvilket
fremgår af datasættet i tabel ??.22 Datasættet indeholder en
række fejl: der er en højde og en diastolisk blodtryksmåling ved
baseline, der er urealistiske, og desuden er værdierne af variablen
køn skrevet på lidt forskellig måde. Endelig skal der udregnes BMI,
som skal bruges til de efterfølgende analyser, og det skal sikres, at kategoriske variable opfattes som faktorer.
Vi bygger dataoprensningen trinvist op. Først bruges filter() til at
beholde individer (rækker i datasættet), der ikke har åbenlyst
forkerte værdier.
DF |> # Tag datasættet og behold rækker,
filter(start>0, # hvor baseline diastolisk er større
hojde>120) # end 0 og højde er større end 120## alder køn diab behandling hojde vaegt start 4 uger
## 1 70 mand nej ny 177 96 99 81
## 2 52 mand nej gammel 180 97 104 93
## 3 46 Mand nej gammel 187 93 94 86
## 4 47 mand nej gammel 185 93 97 91
## 5 74 kvinde nej gammel 162 66 103 90
## 6 68 kvinde nej gammel 161 59 103 90
## 7 56 KVINDE ja ny 168 69 102 81
## 8 59 kvinde ja gammel 167 70 96 86På nuværende tidspunkt er det filtrerede datasæt ikke gemt. Det gøres først til sidst, så det er muligt at se resultaterne undervejs.
mutate() omkoder eksisterende variable eller laver nye. Først laves en ny variabel bmi ud fra vaegt og hojde, og koden til det er
# Definer ny variabel bmi
mutate(bmi = vaegt / (hojde/100)^2)Så tilpasses variablen køn så variablen opfattes som en faktor og så
fejlregistreringer slås sammen. I fct_collapse() specificeres,
hvilke gamle kategorier, der skal samles i nye kategorier. Bemærk at køn overskrives i koden ved at kalde den “nye” variabel det samme navn som den gamle variabel.
# Slå kategorier af køn sammen for at rette indtastningsfejl
# mand samler "mand" og "Mand", mens kvinde samler
# "kvinde" og "KVINDE"
mutate(køn = fct_collapse(køn,
mand = c("mand", "Mand"),
kvinde = c("kvinde", "KVINDE"))) Endelig skal vi sikre, at både diab og behandling opfattes af R som
faktorer. Igen bruges mutate() til at omdefinere og funktionen
factor() omkoder variablen til en faktor. Det er ikke nødvendigt selv at
specificere, hvilke kategorier, der findes, men med argumentet levels kan vælge den interne rækkefølgen på kategorierne, og den første kategori opfattes som referencekategorien.
# Lav diab til en faktor
mutate(diab = factor(diab))
# Lav behandling til et faktor og brug
# kategorien "gammel" som referencekategori
mutate(behandling = factor(behandling,
levels=c("gammel", "ny")))Det hele kan samles til et enkelt kald til mutate().
DF |> # Tag datasættet og behold rækker,
filter(start>0, # hvor baseline diastolisk er større
hojde>120) |> # end 0 og højde er større end 120
mutate(bmi = vaegt / (hojde/100)^2, # Lav BMI
køn = fct_collapse(køn, # Slå grupper sammen
mand = c("mand", "Mand"),
kvinde = c("kvinde", "KVINDE")),
behandling = factor(behandling, # Lav til faktor
levels=c("gammel", "ny")),
diab = factor(diab) # Lav til faktor
)## alder køn diab behandling hojde vaegt start 4 uger bmi
## 1 70 mand nej ny 177 96 99 81 30.64254
## 2 52 mand nej gammel 180 97 104 93 29.93827
## 3 46 mand nej gammel 187 93 94 86 26.59498
## 4 47 mand nej gammel 185 93 97 91 27.17312
## 5 74 kvinde nej gammel 162 66 103 90 25.14861
## 6 68 kvinde nej gammel 161 59 103 90 22.76147
## 7 56 kvinde ja ny 168 69 102 81 24.44728
## 8 59 kvinde ja gammel 167 70 96 86 25.09950Endelig kan vi fjerne de variable, som ikke længere er
nødvendige. select() vælger søjler i datasættet, som skal gemmes,
men med minus foran udvælgelsen angives de søjler, der skal fjernes.
DF |> # Tag datasættet og behold rækker,
filter(start>0, # hvor baseline diastolisk er større
hojde>120) |> # end 0 og højde er større end 120
mutate(bmi = vaegt / (hojde/100)^2, # Lav BMI
køn = fct_collapse(køn, # Slå grupper sammen
mand = c("mand", "Mand"),
kvinde = c("kvinde", "KVINDE")),
behandling = factor(behandling, # Lav til faktor
levels=c("gammel", "ny")),
diab = factor(diab) # Lav til faktor
) |> # Og så
select(-c(hojde, vaegt)) # Fjern hojde/vaegt## alder køn diab behandling start 4 uger bmi
## 1 70 mand nej ny 99 81 30.64254
## 2 52 mand nej gammel 104 93 29.93827
## 3 46 mand nej gammel 94 86 26.59498
## 4 47 mand nej gammel 97 91 27.17312
## 5 74 kvinde nej gammel 103 90 25.14861
## 6 68 kvinde nej gammel 103 90 22.76147
## 7 56 kvinde ja ny 102 81 24.44728
## 8 59 kvinde ja gammel 96 86 25.09950Det oprensede datasæt er stadig ikke gemt. Det gøres ved at gemme resultatet af hele processen i et nyt objekt rettede_data, som så kan bruges til de efterfølgende analyser.
rettede_data <-
DF |> # Tag datasættet og behold rækker,
filter(start>0, # hvor baseline diastolisk er større
hojde>120) |> # end 0 og højde er større end 120
mutate(bmi = vaegt / (hojde/100)^2, # Lav BMI
køn = fct_collapse(køn, # Slå grupper sammen
mand = c("mand", "Mand"),
kvinde = c("kvinde", "KVINDE")),
behandling = factor(behandling, # Lav til faktor
levels=c("gammel", "ny")),
diab = factor(diab) # Lav til faktor
) |> # Og så
select(-c(hojde, vaegt)) # Fjern hojde/vaegttidyverse indeholder to andre nyttige funktioner, summarise() og
group_by(), der henholdsvis bruges til at opsummere data og at splitte
data op i undergrupper for at lave opsummeringer for hver
gruppe. Disse to funktioner introduceres nærmere i kapitel
??.
2.9 Opgaver
Brug R som lommeregner til at udregne følgende:
- \(12\cdot6\)
- \(2\cdot(1+3)^2\)
- \(\log(20) + \sqrt{13}\) [ log henviser til den naturlige logaritme ]
Definer følgende to vektorer
x <- c(1, 3, 6, 2) y <- c(10, 20)Overvej — uden at køre linjerne — hvad følgende kommandoer giver
x-2 x^2 x+y x*y length(x) sum(x) mean(x) sum(x^2)Brug R til at checke, om dine overvejelser var korrekte.
Fødselsvægten (i gram), fostrets alder (i uger) og køn for 8 nyfødte er vist i tabellen nedenfor.
Køn Alder (uge) Fødselsvægt (g) dreng 37 2628 dreng 38 3176 dreng 40 3421 dreng 38 2975 dreng 40 3317 pige 36 2729 pige 40 2935 pige 38 2754 Lav en vektor
vaegt, der indeholder de 8 vægtmålinger. Brug for eksempel funktionenc().Lav to nye vektorer
alderogkon, der indeholder henholdsvis aldersmålingerne og informationen om køn.Udregn en ny vektor,
kg, der indeholder de afrundede vægtmålinger i kg. Brug funktionenround()til at afrunde fødselsvægten til nærmeste 100 gram. Brug hjælpesiden tilround()til at finde ud af, hvordan man vælger antallet af betydende cifre, når man afrunder.23De nyfødte piger svarer til måling nummer 6, 7 og 8. Brug indicering af vektoren
vaegttil at udtrække fødselsvægten for observation 6, 7 og 8. Hvad er den gennemsnitlige fødselsvægt for de tre piger?Det er sjældent, at man kender de specifikke observationsnumre, man vil udtrække. Brug i stedet oplysningerne fra vektoren
kontil at udtrække fødselsvægten for pigerne.Erstat det 4. element af
vaegtiDFfra 2975 til værdien 3075.Udregn den gennemsnitlige fødselsvægt for alle 8 personer i
DF.24Lav en data frame,
DF, der indeholder de 4 variablekon,alder,vaegtogkg. Prøv at køre kommandoenhead(DF)Hvad viser den?
Se hjælpesiden til
head()for at se, hvordan man ændrer antallet af linjer, der vises.25Prøv at køre kommandoen
summary(DF)Hvad viser den? Hvorfor er formatet forskelligt for
vaegtogkon?
Datasættet
morbarnfra pakkenstat4medindeholder oplysninger om en tilfældig stikprøve på 400 førstegangsfødende mødre fra det såkaldte Mor-Barn studie XXX (indsæt ref), der følger levendefødte børn fra terminsugerne 37-42, og hvor moderen ikke drak under graviditeten. Datasættet indeholder seks registreringer om hver moder: alder (i år), ryger (ja/nej), kaffedrikker (ja/nej), uge (gestationsalder ved fødslen i uger), vaegt (barnets vægt i gram) og længde (barnets længde i cm).Data kan indlæses ved at skrive kommandoen
library("stat4med") # Indlæs pakken data("morbarn") # Gør data tilgængeligeDe efterfølgende spørgsmål kan med fordel besvares ved brug af funktionerne fra
tidyversepakken.- Hvad er gennemsnitsalderen for de 400 førstegangsfødende? Hvad er gennemsnitsvægten for de nyfødte?
- Lav en ny data frame kaldet
deldata, der kun indeholder de mødre, der hverken ryger eller drikker kaffe.26 - Lav fødselsvægt defineres ofte som børn, der har en fødselsvægt mellem 1500 til 2500 gram. Lav en ny variabel,
lavvaegt, der erTRUE, hvis børnene har lav fødselsvægt, ogFALSEellers. - Hvor mange børn har lav fødselsvægt?27
Hver gang man starter på et nyt projekt er det en god ide at lave et bibliotek/en folder på computeren, der indeholder alle data og R scripts. Det sikrer, at man har data og programmer samlet, og at man ikke får sammenblandet filer fra forskellige analyser.
Når R starter op starter den typisk i samme bibliotek, og det kan være nødvendigt at sætte det bibliotek, hvor den skal finde og gemme filer.
- Brug funktionen
getwd()(get working directory) til at se, hvilket bibliotek R leder efter filer i. - Opret et ny bibliotek,
testRpå computeren. Brug stifinderen til at gøre dette. - Sæt det nye arbejdsbibliotek til
testR. Dette kan gøres i RStudio via menupunktet Session -> Set Working Directory -> Choose Directory. Et alternativ er at bruge funktionensetwd(). - Kør
getwd()igen for at checke at det nye arbejdsbibliotek ertestR.
- Brug funktionen
Tabeller. Lav en matrix …
Import af data. CSV? Excel? Ha’ en excel-fil på hjemmesiden. Læs den ind, gem som csv. Prøv at læse ind via …
R kan hentes fra
www.r-project.org. Fra hjemmesiden vælges først download (CRAN), derefter en server, hvorefter R kan hentes og installeres.↩︎RStudios hjemmeside er
www.rstudio.comog herfra kan man downloade RStudio.↩︎Kommandoerne i RStudios editor kan gemmes som dokumentation for de analyser, der er lavet. Filer med kommandoer til R har typisk efternavnet
.Reller.r, men det er bare helt almindelige tekstfiler. Det anbefales at skrive alle kommandoerne i editoren, da det sikrer, at det er muligt at reproducere og genskabe alle resultater.↩︎Opgave XXX viser, hvordan arbejdsbiblioteket skiftes.↩︎
Skrives navnet på en funktion uden at angive parenteser så vises funktionens kildekode. Det gør det muligt direkte at se, hvad funktionerne i R faktisk gør.↩︎
install.packages()svarer til, at installere en lampe i et hus — det skal kun gøres en gang.library()svarer til at tænde på kontakten, og det skal man huske at gøre, hver gang man starter et program og har “brug for lyset”.↩︎Foran resultatet står et
[1], som vi kan ignorere indtil videre. Forklaringen kommer i afsnit 2.4.↩︎Pilen består af to tegn:
<(mindre end) og-(minus). Pilen<-kan opfattes som om, at man sender resultatet over i det valgte objekt.↩︎På samme vis er funktionen
log()ikke det samme som funktionenLOG()ellerLog().↩︎Desuden er internettet er en rig ressource til at søge efter hjælp til R. Der er tusindevis af sider med hjælp, eksempler, forklaringer og videoer.↩︎
Body-mass index er defineret som en persons vægt (målt i kg) delt med højde (målt i meter) kvadreret.↩︎
Hvis længden af den korte vektor ikke går op i længden af den lange vektor så bliver den korte vektor stadig gentaget indtil den matcher længden af den lange, men en advarsel skrives på skærmen.↩︎
DF$v4læses som “variabelv4fraDF”, og den adskiller sig fraDF2$v4, der er en helt anden vektor (dog med samme navn), og som findes i data framenDF2.↩︎Alternativt skal længden på den korte vektor gå op i længden på de øvrige variable i data framen, så den kan gentages til at have samme længde.↩︎
Data frames og matricer er begge rektangulære strukturer, men matricer kan kun indeholde tal. I en matrix er der til gengæld ikke nogen typeforskel på rækker og søjler.↩︎
Med argumentet
byrow=TRUEtilmatrix()fyldes matricen op rækkevis i stedet for søjlevis.↩︎Filen
forsøgsdata.xlsxantages at ligge i samme bibliotek som R sessionen. Ellers skal den fulde sti til filen skrives. Filenforsøgsdata.xlsxkan hentes fra bogens hjemmeside.↩︎En CSV-fil (comma-separated values) er en almindelig tekstfil, hvor søjlerne i datasættet er adskilt med comma, semikolon eller mellemrum. En fil med dette indhold findes på bogens hjemmeside.↩︎
Dette er specielt vigtigt for at kunne reproducere resultaterne. Hvis ikke de præcise argumenter til indlæsning bliver gemt, så risikerer det, at filen senere indlæses på en anden måde, hvilket kan give andre resultater. ↩︎
tidyversebruger en udvidet data frame kaldettibblesitidyverse, men den fungerer som en data frame. Udtales “tibbles” lidt hurtigt lyder det næsten ligesom “tables”.↩︎|>består af to tegn: “lodret streg” og “større end” og kaldes en “pipe”, da den som et rør fører resultatet fra en funktion videre til den næste funktion.↩︎Datasættet findes også i pakken stat4med og hedder XXX.↩︎
Fif:
round()afrunder betydende cifre efter kommaet. For at afrunde til nærmeste 100 g kan det derfor være nødvendigt først at dividere den oprindelig vægt, dernæst afrunde og så gange op igen.↩︎Brug enten
$eller indicering til at trække den relevante variabel ud af data framen.↩︎head()er særlig nyttig, hvis man vil have et hurtigt overblik over indholdet i en data frame eller en vektor.↩︎Husk, at man skal bruge to lighedstegn, når man checker om en variabel er lig en bestemt værdi.↩︎
Man kan bruge funktionen
sum()til at tælle op, hvor oftelavvaegterTRUE.↩︎